112學年度陽明交通大學生物科技學院碩士班考試入學備取通知
112學年度碩士班考試入學備取生名單及報到注意事項
即日起至112年5月17日(三)下午3點前 (不含六、日、國定假日)完成報到手續
一、報到地點:新竹市博愛街75號賢齊館325室。
二、報到登記時需攜帶以下資料:大學成績單正本、畢業證書正本(如為應屆畢業生,請填寫切結書)
※未報到者視同放棄,缺額將由備取生依序遞補登記報到。
如欲放棄請填寫回覆放棄聲明書或回覆信件告知。
生科系
4600005備16
112學年度陽明交通大學生物科技學院碩士班考試入學備取通知
112學年度碩士班考試入學備取生名單及報到注意事項
即日起至112年5月17日(三)下午3點前 (不含六、日、國定假日)完成報到手續
一、報到地點:新竹市博愛街75號賢齊館325室。
二、報到登記時需攜帶以下資料:大學成績單正本、畢業證書正本(如為應屆畢業生,請填寫切結書)
※未報到者視同放棄,缺額將由備取生依序遞補登記報到。
如欲放棄請填寫回覆放棄聲明書或回覆信件告知。
生科系
4600005備16
1. 於資格考口試一週前填寫資格考學位考試申請表,並將考試申請表擲交系辦相關事務負責人。
註1:博士班口試委員5-9位(含指導教授,不限校內外,學校僅付最多三位校外委員,若超過三位校外委員及校內委員請自行支付口試委員費用)
註2:若您論文有涉及人體試驗、動物實驗,敬請檢附相關證明文件。
2. 口試前請先查詢是否以符合下列事項:(請配合博士班研究生入學與修讀辦法)
(1)候選人當於考試前修滿9學分(含)以上之課程,其成績必需在70分以上。
(2)必需已通過二學期博士班書報討論。
(3)繳交博士論文研究計畫及初步成果之書面報告,或加上其他成為候選人值得參考之書面資料。
(4)一般生研究生第四年結束前(休學不計入年限)需發表SCI論文總點數2.0以上(含)或SCI領域學門在前20%之論文一篇;在職生第六年結束前(休學不計入年限)需發表SCI論文總點數2.0以上(含)或SCI領域學門在前20%之論文一篇。
3.口試當天請先列印口試委員評分表、口試評分總表及口試審定書
4.博士候選人資格考口試委員會審定書一份(依照國立陽明交通大學學位論文格式規範)請將您的資料填妥(打字),於口試當天請學位考試委員簽名。
5.口試結束後請口試召集人將評分表及審定書送至系辦。
相關資料下載
1.推薦書
(1)分醫所:
(2)生科系:
(3)生資所:
(4)生醫工博學程:
(5)產業博士班:
(6)跨領域神經學程:
王雲銘教授研究團隊發表研究成果於 Sensors and Actuators B: Chemical
連結網址:
https://www.sciencedirect.com/science/article/pii/S0925400523006755
Abstract
The COVID-19 pandemic has become a global catastrophe, affecting the health and economy of the human community. It is required to mitigate the impact of pandemics by developing rapid molecular diagnostics for SARS-CoV-2 virus detection. In this context, developing a rapid point-of-care (POC) diagnostic test is a holistic approach to the prevention of COVID-19. In this context, this study aims at presenting a real-time, biosensor chip for improved molecular diagnostics including recombinant SARS-CoV-2 spike glycoprotein and SARS-CoV-2 pseudovirus detection based on one-step-one-pot hydrothermally derived CoFeBDCNH2-CoFe2O4 MOF-nanohybrids. This study was tested on a PalmSens-EmStat Go POC device, showing a limit of detection (LOD) for recombinant SARS-CoV-2 spike glycoprotein of 6.68 fg/mL and 6.20 fg/mL in buffer and 10% serum-containing media, respectively. To validate virus detection in the POC platform, an electrochemical instrument (CHI6116E) was used to perform dose dependent studies under similar experimental conditions to the handheld device. The results obtained from these studies were comparable indicating the capability and high detection electrochemical performance of MOF nanocomposite derived from one-step-one-pot hydrothermal synthesis for SARS-CoV-2 detection for the first time. Further, the performance of the sensor was tested in the presence of Omicron BA.2 and wild-type D614G pseudoviruses.
親愛的同學們,您好:
恭喜您們即將畢業
雖然在畢業前您們有很多很急的事情需要處理,但下列事項有關您的權益,請您花點時間看看
1.於畢業論文口試兩週前(因學校公文往返申請學位考試委員聘書及口試費用需兩週的時間,學校規定公文未完成前口試屬無效,請各位同學須特別留意)填寫學位考試申請表,並將學位考試申請表及論文原創性比對相關證明擲交系辦相關事務負責人。
註1:碩士班口試委員3-5位(含指導教授【支給論文指導費,不再支給委員口試費】,但學校僅最多支付三位委員費用,若超過三位由實驗室自行支付口試委員費用)
註2:若您論文有涉及人體試驗、動物實驗,敬請檢附相關證明文件。
2.登記借用學位考試場地(請自行於學位考試場地外登記)。
3. 口試以公開舉行為原則,須於事前於系辦登錄口試時間、地點及論文題目。
4.口試當天請先列印口試委員評分表、口試評分總表、成績資料表及口試審定書,並於當天跟系辦領取校外委員口試費用領據及貴賓停車證
5.畢業論文口試委員會審定書一份,(依照國立陽明交通大學學位論文格式規範)請將你的資料填妥(打字),於口試當天請學位考試委員簽名,以便裝訂於論文前頁,並將正本裝訂於繳交給學校圖書館的論文中。(註:請留意所名的正確性)(生科院下所名:生物科技學系(生科系所合一請勿自行加上研究所)、分子醫學與生物工程研究所、生物資訊及系統生物研究所、生醫科學與工程博士學位學程、跨領域神經科學博士學位學程、生物科技學院產業博士班。)不同所別有不同的審定書請多留意,務必列印您所屬系所的使用版本,且口試委員部分處請空白留給口試委員簽名切勿打字)
6.口試結束後請口試召集人將評分表送至系辦,同學們請將【領據簽單】、【成績資料表】送回系辦。「學位考試成績資料表」第一學期需於1月31日前繳交;第二學期需於7月31日前繳交。
7. 碩士學位論文(含摘要)以中文或英文撰寫為原則,並須符合本校學位論文格式規範。請上圖書館國立陽明交通大學博碩士論文全文系統將學位論文書目摘要以及全文電子檔上網建檔。論文審核通過後,需繳交論文三冊(一冊本校圖書館陳列,一冊由國家圖書館收藏,一冊由所上收藏)。紙本論文除涉及機密、專利事項或依法不得提供者得提出相關證明文件至系辦申請延後公開外,否則紙本論文不得延後公開。(https://idm.nycu.ust.edu.tw/sso/886UST_NYCU/oidc/login/?next=/openid/886UST_NYCU/authorize%3Fclient_id%3D702412%26redirect_uri%3Dhttps%3A//etd.lib.nctu.edu.tw/cgi-bin/gs32/tugsweb.cgi/ccd%3D_6w0Zl/idmdispatch%3F%26response_type%3Dcode%26scope%3Dopenid)
註:以圖書館借還書帳號密碼登入
8.需列印授權書依序將授權書裝訂於論文中。
9.各實驗室財產管理者,需先填寫財產移交清冊,於離校時一併繳交至系辦。
10. 進入校園單一入口(https://portal.nycu.edu.tw/#/login?redirect=%2F)啟動交大校區離校簽核系統,依序請指導教授簽核、繳交一本論文至系辦後系辦簽核、至圖書館繳交一本論文、職涯發展組(線上審核)、駐警隊(線上審核)、最後系統通知請持學生證及論文紙本1本至註冊組(科學一館1樓)領取證書。
11.學位考試通過後,「學位考試成績資料表」若第一學期需於1月31日前繳交;第二學期需於7月31日前繳交給註冊組。學位論文紙本之繳交期限為舉行學位考試日的次學期開學前最後一個工作日,逾期未繳交論文紙本且未達修業年限者,次學期仍應註冊。
12.修業年限屆滿者,未於年限屆滿當學期繳交學位考試成績資料表或未於次學期開學前最後一個工作日前繳交紙本論文,應予退學。
13.系所館舍門卡使用期限至離校手續辦妥隔天為止。
14.國立陽明交通大學畢業生學位證書發給作業要點,請參考本校註冊組網頁。
15.國立陽明交通大學學位論文格式規範,請參考本校註冊組網頁。封面顏色請先洽系辦查詢(生科系為綠色、分醫所為黃色、生資所為藍色)。
最後再次恭喜畢業
以上程序若有疑問
歡迎洽詢系辦羅悅瑜分機56901
親愛的同學們,您好:
恭喜您們即將畢業
雖然在畢業前您們有很多很急的事情需要處理,但下列事項有關您的權益,請您花點時間看看
1.畢業前請先查詢是否以符合下列事項:(請配合博士班研究生入學與修讀辦法)
2.於畢業論文口試兩週前(因學校公文往返申請學位考試委員聘書及口試費用需兩週的時間)填寫學位考試申請表,並將學位考試申請表及論文原創性比對證明、發表論文第一頁等擲交系辦相關事務負責人。
註1:博士班口試委員5-9位(含指導教授【僅支給論文指導費,不再支給委員口試費】,但學校僅最多支付七位委員費用,若超過七位由實驗室自行支付口試委員費用,校內外委員各需超過1/3)
註2:若您論文有涉及人體試驗、動物實驗,敬請檢附相關證明文件。
3.登記借用學位考試場地(請自行於學位考試場地外登記)。
4. 口試以公開舉行為原則,須於事前於系辦登錄口試時間、地點及論文題目。
5.口試當天請先列印口試委員評分表、口試評分總表、學位考試成績資料表及口試審定書,並於當天跟系辦領取校外委員口試費用領據及貴賓停車證。
6.畢業論文口試委員會審定書一份(依照國立陽明交通大學學位論文格式規範)請將你的資料填妥(打字),於口試當天請學位考試委員簽名,以便裝訂於論文前頁,並將正本裝訂於繳交給學校圖書館的論文中。(註:請留意所名的正確性)(生科院下所名:生物科技學系(生科系所合一請勿自行加上研究所)、分子醫學與生物工程研究所、生物資訊及系統生物研究所、生醫科學與工程博士學位學程、跨領域神經科學博士學位學程、生物科技學院產業博士班。)不同所別有不同的審定書請多留意,務必列印您所屬系所的使用版本,且口試委員部分處請空白留給口試委員簽名切勿打字)
7. 口試結束後請口試召集人將評分表送至系辦,同學們請將【領據簽單】、【成績資料表】送回系辦。「學位考試成績資料表」第一學期需於1月31日前繳交;第二學期需於7月31日前繳交。
8. 博士學位論文(含摘要)以中文或英文撰寫為原則,並須符合本校學位論文格式規範。請上圖書館國立陽明交通大學博碩士論文全文系統將學位論文書目摘要以及全文電子檔上網建檔。論文審核通過後,需繳交論文三冊(一冊本校圖書館陳列,一冊由國家圖書館收藏,一冊由所上收藏)。紙本論文除涉及機密、專利事項或依法不得提供者得提出相關證明文件至系辦申請延後公開外,否則紙本論文不得延後公開。(https://idm.nycu.ust.edu.tw/sso/886UST_NYCU/oidc/login/?next=/openid/886UST_NYCU/authorize%3Fclient_id%3D702412%26redirect_uri%3Dhttps%3A//etd.lib.nctu.edu.tw/cgi-bin/gs32/tugsweb.cgi/ccd%3D_6w0Zl/idmdispatch%3F%26response_type%3Dcode%26scope%3Dopenid)
註:以圖書館借還書帳號密碼登入
9. 進入校園單一入口(https://portal.nycu.edu.tw/#/login?redirect=%2F)啟動交大校區離校簽核系統,依序請指導教授簽核、繳交一本論文至系辦後系辦簽核、至圖書館繳交一本論文、職涯發展組(線上審核)、駐警隊(線上審核)、最後系統通知請持學生證及論文紙本1本至註冊組(科學一館1樓)領取證書。
11.學位考試通過後,「學位考試成績資料表」若第一學期需於1月31日前繳交;第二學期需於7月31日前繳交給註冊組。學位論文紙本之繳交期限為舉行學位考試日的次學期開學前最後一個工作日,逾期未繳交論文紙本且未達修業年限者,次學期仍應註冊。
12.修業年限屆滿者,未於年限屆滿當學期繳交學位考試成績資料表或未於次學期開學前最後一個工作日前繳交紙本論文,應予退學。
13.系所館舍門卡使用期限至離校手續辦妥隔天為止。
14.國立交通大學學位論文格式規範,請參考本校註冊組網頁。封面顏色為深紅色,樣本請先洽系辦查詢。論文裝訂需採精裝。
最後再次恭喜畢業
以上程序若有疑問
歡迎洽詢系辦羅悅瑜分機56901
學位考試申請表
國立陽明交通大學生物科技學院
112學年度第一學期逕行修讀博士學位開始辦理申請
(111年5月8日公佈)
一、申請資格:
(一)修讀學士學位應屆畢業生,修業期間成績優異,並具有研究潛力。
(二)修讀碩士學位學生,修業期間成績優異,並具有研究潛力。
二、報名日期:112年5月15日起至112年6月30日截止。
三、繳交文件:
(一)申請書
(二)助理教授以上推薦函兩份以上,其中一份需由指導教授、系主任或所長推薦。
(三)歷年成績單(含各學期名次證明)。
(四)成果摘要(至多A4紙5頁)。
(五) 研究計畫(至多A4紙5頁)。
※上述1-5項資料於報名時繳交至系辦,並註明報考所別,否則無法受理。
(六)口試當天繳交口試報告摘要。
四、逕行修讀博士錄取名額:
生物科技學系5名
生物資訊及系統生物研究所2名
分子醫學與生物工程研究所1名
生物科技學院產學博士班1名
生醫科學與工程博士學位學程2名
五、評分標準:
(一)審查佔50﹪
(二)口試佔50﹪(其中過去成果佔80﹪,未來研究佔20﹪)
六、口試時間:112年7月11日(二)
七、口試地點:博愛校區賢齊館3樓
112學年度碩士班考試入學備取生名單及報到注意事項
即日起至112年5月11日(四)下午3點前 (不含六、日、國定假日)完成報到手續
一、報到地點:新竹市博愛街75號賢齊館325室。
二、報到登記時需攜帶以下資料:大學成績單正本、畢業證書正本(如為應屆畢業生,請填寫切結書)
※未報到者視同放棄,缺額將由備取生依序遞補登記報到。
如欲放棄請填寫回覆放棄聲明書或回覆信件告知。
生科系
4600017備14
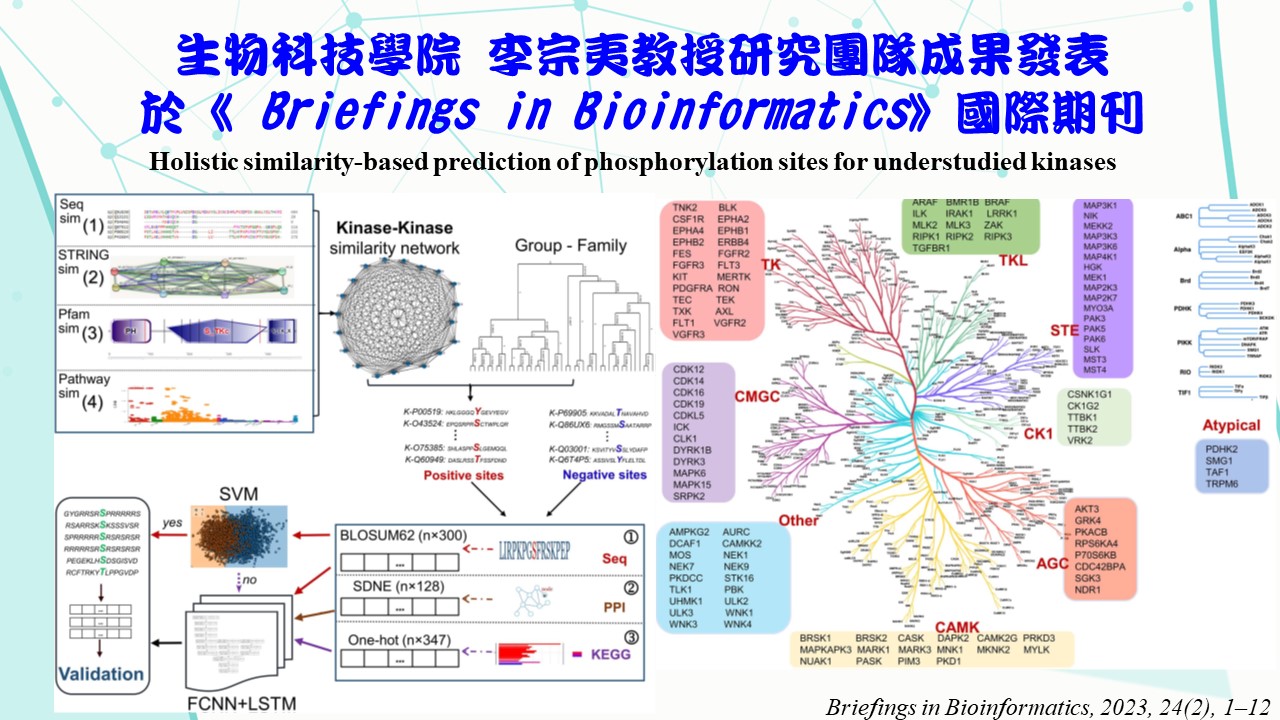
李宗夷教授研究團隊發表研究成果於Briefings in Bioinformatics
連結網址:https://pubmed.ncbi.nlm.nih.gov/36810579/
Phosphorylation is an essential mechanism for regulating protein activities. Determining kinase-specific phosphorylation sites by experiments involves time- consuming and expensive analyzes. Although several studies proposed computational methods to model kinase-specific phosphorylation sites, they typically required abundant experimentally verified phosphorylation sites to yield reliable predictions. Nevertheless, the number of experimentally verified phosphorylation sites for most kinases is relatively small, and the targeting phosphorylation sites are still unidentified for some kinases. In fact, there is little research related to these understudied kinases in the literature. Thus, this study aims to create predictive models for these understudied kinases. A kinase-kinase similarity network was generated by merging
the sequence-, functional-, protein-domain- and ‘STRING’-related similarities. Thus, besides sequence data, protein-protein interactions and functional pathways were also considered to aid predictive modelling. This similarity network was then integrated with a classification of kinase groups to yield highly similar kinases to a specific understudied type of kinase. Their experimentally verified phosphorylation sites were leveraged as positive sites to train predictive models. The experimentally verified phosphorylation sites of the understudied kinase were used for validation. Results demonstrate that 82 out of 116 understudied kinases were predicted with adequate performance via the proposed modelling strategy, achieving a balanced accuracy of 0.81, 0.78, 0.84, 0.84, 0.85, 0.82, 0.90, 0.82 and 0.85, for the ‘TK’, ‘Other’, ‘STE’, ‘CAMK’, ‘TKL’, ‘CMGC’, ‘AGC’, ‘CK1’ and ‘Atypical’ groups, respectively. Therefore, this study demonstrates that web-like predictive networks can reliably capture the underlying patterns in such understudied kinases by harnessing relevant sources of similarities to predict their specific phosphorylation sites.
何信瑩教授研究團隊發表研究成果於Human Genetics and Genomics Advances
連結網址:https://pubmed.ncbi.nlm.nih.gov/37124139/
The ability to detect cancer at an early stage in patients who would benefit from effective therapy is a key factor in increasing survivability. This work proposes an evolutionary supervised learning method called CancerSig to identify cancer stage-specific microRNA (miRNA) signatures for early cancer predictions. CancerSig established a compact panel of miRNA signatures as potential markers from 4,667 patients with 15 different types of cancers for the cancer stage prediction, and achieved a mean performance: 10-fold cross-validation accuracy, sensitivity, specificity, and area under the receiver operating characteristic curve of 84.27% ± 6.31%, 0.81 ± 0.12, 0.80 ± 0.10, and 0.80 ± 0.06, respectively. The pan-cancer analysis of miRNA signatures suggested that three miRNAs, hsa-let-7i-3p, hsa-miR-362-3p, and hsa-miR-3651, contributed significantly toward stage prediction across 8 cancers, and each of the 67 miRNAs of the panel was a biomarker of stage prediction in more than one cancer. CancerSig may serve as the basis for cancer screening and therapeutic selection.
李宗夷教授研究團隊發表研究成果於Microbiology Spectrum
連結網址:https://pubmed.ncbi.nlm.nih.gov/37042778/
In clinical microbiology, matrix-assisted laser desorption ionization-time-of-flight mass spectrometry (MALDI-TOF MS) is frequently employed for rapid microbial identification. However, rapid identification of antimicrobial resistance (AMR) in Escherichia coli based on a large amount of MALDI-TOF MS data has not yet been reported. This may be because building a prediction model to cover all E. coli isolates would be challenging given the high diversity of the E. coli population. This study aimed to develop a MALDI-TOF MS-based, data-driven, two-stage framework for characterizing different AMRs in E. coli. Specifically, amoxicillin (AMC), ceftazidime (CAZ), ciprofloxacin (CIP), ceftriaxone (CRO), and cefuroxime (CXM) were used. In the first stage, we split the data into two groups based on informative peaks according to the importance of the random forest. In the second stage, prediction models were constructed using four different machine learning algorithms-logistic regression, support vector machine, random forest, and extreme gradient boosting (XGBoost). The findings demonstrate that XGBoost outperformed the other four machine learning models. The values of the area under the receiver operating characteristic curve were 0.62, 0.72, 0.87, 0.72, and 0.72 for AMC, CAZ, CIP, CRO, and CXM, respectively. This implies that a data-driven, two-stage framework could improve accuracy by approximately 2.8%. As a result, we developed AMR prediction models for E. coli using a data-driven two-stage framework, which is promising for assisting physicians in making decisions. Further, the analysis of informative peaks in future studies could potentially reveal new insights. IMPORTANCE Based on a large amount of matrix-assisted laser desorption ionization-time-of-flight mass spectrometry (MALDI-TOF MS) clinical data, comprising 37,918 Escherichia coli isolates, a data-driven two-stage framework was established to evaluate the antimicrobial resistance of E. coli. Five antibiotics, including amoxicillin (AMC), ceftazidime (CAZ), ciprofloxacin (CIP), ceftriaxone (CRO), and cefuroxime (CXM), were considered for the two-stage model training, and the values of the area under the receiver operating characteristic curve (AUC) were 0.62 for AMC, 0.72 for CAZ, 0.87 for CIP, 0.72 for CRO, and 0.72 for CXM. Further investigations revealed that the informative peak m/z 9714 appeared with some important peaks at m/z 6809, m/z 7650, m/z 10534, and m/z 11783 for CIP and at m/z 6809, m/z 10475, and m/z 8447 for CAZ, CRO, and CXM. This framework has the potential to improve the accuracy by approximately 2.8%, indicating a promising potential for further research.